Erst am vergangenen Sonntag (10. Dezember) hat "Doom" seinen 30. Geburtstag gefeiert. Was das Spiel, welches das Genre der Egoshooter entscheidend prägte, bis heute am Leben erhält, sind aber nicht nur seine direkten Nachfolger, sondern auch eine treue Fangemeinde, die versucht, den Klassiker auf allen erdenklichen Geräten laufen zu lassen.
Das geschieht aber nicht immer nur aus purem Vergnügen am Basteln, sondern durchaus auch mit wissenschaftlichem Hintergrund. So auch das Projekt von Lauren Ramlan, PhD-Studentin am renommierten Massachusetts Institute of Technology (MIT). Sie hat nun eine neue Unterlage für das Game gefunden, nämlich Escherichia-coli-Bakterien, besser bekannt unter dem verkürzten Namen E. coli.
Die Vielseitigkeit dieser eigentlich aus dem Darm stammenden Bakterienart macht sie beliebt für Experimente aller Art. Im vorliegenden Fall dienten sie nicht zur Berechnung des Spiels, sondern als Bildschirm. Auch das, so merkt Ramlan in ihrer Projektbeschreibung korrekt an, gilt im Geiste der "Doom Runs On Everything"-Mission gemeinhin als "zum Laufen gebracht". Den dazugehörigen Quellcode hat sie auf Colab veröffentlicht.
Could you run Doom on cells?
Fluoreszierende Zellen
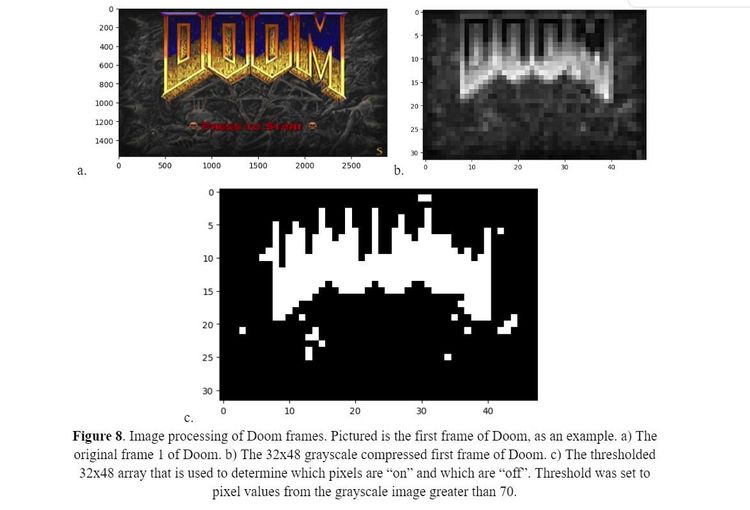
Die Zellen wurden für diesen Versuch mittels Computermodell simuliert – was sehr akkurat möglich ist – und in ein Gitter mit 48 mal 32 Blöcken angeordnet. Jeder Block dient als Pixel, die Darstellung erfolgt in zwei Farben. Die Zellen werden, stark vereinfacht erklärt, durch die Manipulation ihres Erbträgers (Plasmid) bei Bedarf mittels zweier interagierender genetischer Schaltkreise zum Fluoreszieren gebracht
Um festzulegen, welche Zellen jeweils aufleuchten, wird die Darstellung von "Doom" in Graustufen konvertiert und jedes Bild ausgewertet. Zellen, deren korrespondierende Pixel einen Wert von 70 überschreiten, werden zum Leuchten angeregt.
Die Framerate ist allerdings noch etwas entfernt von guter Spielbarkeit. Es dauert rund 70 Minuten, bis die angesteuerten Zellen die beste Darstellung am Gitter erzielen und insgesamt etwa acht Stunden und 20 Minuten, bis sie wieder ihren Ursprungszustand einnehmen.
"Wenn 'Doom' und das Leben eins werden"
Dementsprechend würde ein Durchspielen von "Doom" auf diesem Zellbildschirm zu einem Generationenprojekt. Ausgehend von einem Zeitaufwand von fünf Stunden für ein durchschnittliches Playthrough (basierend auf Zahlen der Website "How Long To Beat“) sowie der maximalen Darstellungsrate von 35 Bildern pro Sekunde der originalen „Doom"-Engine wären dafür 599 Jahre zu veranschlagen. Oder wie Ramlan es formuliert: "Mit Utopien aus synthetischer Biologie am Horizont sind wir nur ein halbes Jahrtausend von der bisher größten Errungenschaft entfernt: wenn 'Doom' und das Leben eins werden."
Die Forscherin hat bereits Ideen verfasst, wie sich dieser Zeitbedarf potenziell deutlich verkürzen ließe. So schlägt sie etwa einen Löschmechanismus vor, der das Abklingen der Fluoreszenz beschleunigen könnte. Eine weitere Ergänzung ist ein an einen Zwischenspeicher gekoppeltes Voraussagesystem, um Zellen weiter leuchten zu lassen, die auch in den folgenden Frames ohnehin aktiviert würden. Ebenso ließe sich ein Mindestniveau des Fluoreszenzlevels ermitteln, ab dessen Unterschreitung man bereits das nächste Bild ins System einspeisen kann. (gpi, 12.12.2023)