Damit eine menschliche Zelle einen bestimmten Lebensweg einschlagen kann und beispielsweise zu einer Muskel-, Nerven-, Haut- oder Darmzelle wird, müssen viele Gene präzise gesteuert werden. Dafür gibt es Ableseverstärker, sogenannte Enhancer. Diese speziellen DNA-Abschnitte sind dazu in der Lage, Gene ein- und auszuschalten.
Wiener Biologinnen und Biologen ist es mithilfe künstlicher Intelligenz gelungen, den DNA-Code dieser Enhancer zu knacken. Die neuen Erkenntnisse ermöglichen es dem Team um Alexander Stark vom Institut für Molekulare Pathologie (IMP) in Wien nun, die Aktivität der Gen-Ableseverstärker in den Körperteilen vorherzusagen, die Folgen von Mutationen abzuschätzen und synthetische Enhancer zu erzeugen.
Unbekannte Enhancer
"Jede Zelle im menschlichen Körper enthält dieselben Gene, aber verschiedene Gewebe aktivieren unterschiedliche Gruppen von Genen", erklärten die Forschenden. Von den für diese Prozesse so wichtigen Enhancern wusste man bisher zwar, dass sie Gene steuern – nicht aber, wie diese regulatorische Funktion in der DNA hinterlegt ist. Während die ersten Enhancer bereits in den frühen 1980er-Jahren entdeckt wurden, haben Wissenschafterinnen und Wissenschafter erst im letzten Jahrzehnt Methoden entwickelt, um Enhancer experimentell zu identifizieren.
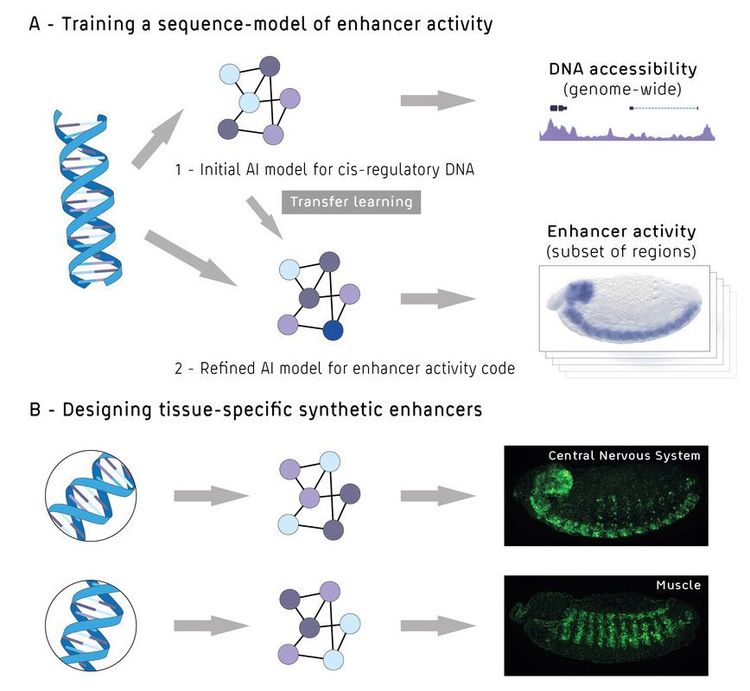
Darauf aufbauend hatten sich die Forschenden um Stark unter anderem vorgenommen, die Aktivität von Enhancern aus ihrer DNA-Sequenz zu entschlüsseln. Jüngste Fortschritte in der Genomik und der künstlichen Intelligenz erlaubten neue Ansätze, den Enhancer-Code zu knacken. Die Gruppe entwickelten ein leistungsfähiges Deep and Transfer Learning Modell, das sie mit großen Datensätzen der Fruchtfliege Drosophila melanogaster trainierten, einem wichtigen Modellorganismus der Entwicklungsbiologie.
Training an der Fruchtfliege
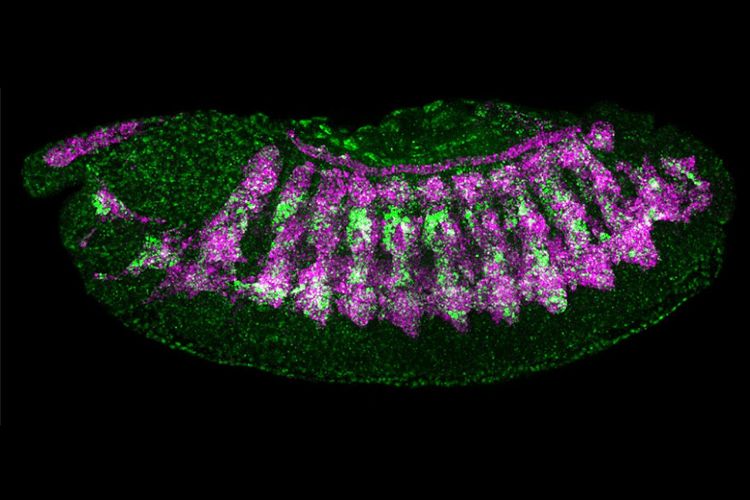
Zunächst wurde das Modell an genomweiten DNA-Sequenzen und entsprechenden Daten der DNA-Zugänglichkeit im Chromatin trainiert, einem relativ leicht messbaren Vorhersagekriterium für Enhancer. Basierend auf diesem ersten Modell wurde ein zweites trainiert, das DNA-Sequenzen direkt mit spezifischen Enhancer-Aktivitäten verknüpfte. Das Modell war nun in der Lage, die Enhancer-Aktivität für fünf Gewebearten in Fruchtfliegenembryonen vorherzusagen: das zentrale Nervensystem, den Teilbereich des Gehirns, Epidermis, Darm und Muskeln.
In einem nächsten Schritt kehrten die Forschenden in das Labor zurück: Mit etablierten molekularbiologischen Methoden schufen sie 40 synthetische Enhancer, die zuvor am Computer rechnerisch entwickelt wurden, und testeten sie in lebenden Embryos von Fruchtfliegen. Tatsächlich aktivierten die Enhancer Gene in den Zielgeweben.
"Die Möglichkeit, künstliche Enhancer mit spezifischen Eigenschaften zu bauen, eröffnet ungeahnte Möglichkeiten für die gezielte Steuerung von Genexpression", sagte Bernardo de Almeida, Erstautor der im Fachjournal "Nature" veröffentlichten Studie. Zukünftige Anwendungen könnten in der synthetischen Biologie oder Gentherapie liegen, wo die präzise Gestaltung und Manipulation von Genexpressionsmustern eine Voraussetzung ist. (red, 12.12.2023)